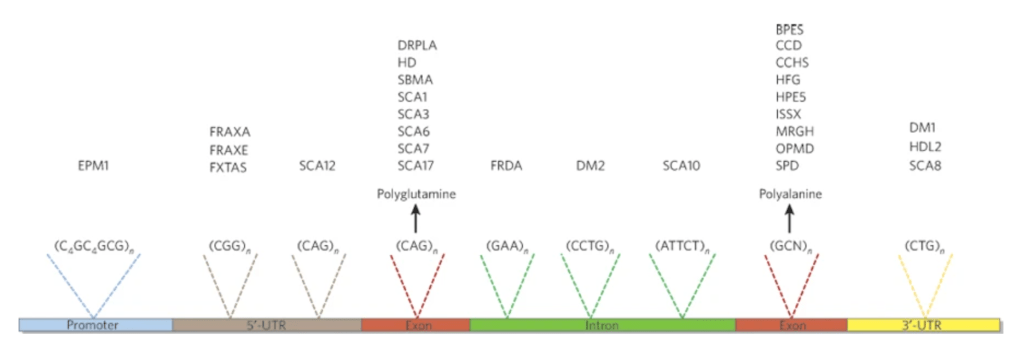
前面有稍微分享一下2021 臨床次世代定序實務與應用概論這堂課的內容,當然打鐵趁熱也針對一些主題,自己做了些延伸和討論,這邊就針對CAP和台灣的指引來分享一下,將次世代定序使用在臨床所牽涉到實驗室端的細節是以前沒有面對過的,相對於傳統實驗室檢驗單項指標,次世代定序從檢測原理到結果都是高通量的輸出,也許用臨床檢查來譬喻有點像是病人基因的電腦斷層,但是更為複雜幾倍。
大概7-8年前,世界各國基因體學相關的臨床學術組織便開始努力制定相對應的實驗室指引來幫忙導入次世代定序使用,同時也有如實驗室開發檢驗指引等法規來解套基因定序作為檢驗項目的醫療法規問題,另外,廠商也推動一些機型的法規認證。

在2013年illumina公司的MiSeqDx平台獲得美國FDA的核准,是第一台獲得IVD認證的機台,同時其三個試劑分別是MiSeqDx universal kit和兩個跟囊腫性纖維化(Cystic fibrosis)的定序同時取得認證。後續Ion PGM Dx system也在2017年6月22號成為美國第二台FDA認證的醫學檢驗用NGS儀器。
而後續產業界跟學術界慢慢希望可以用實驗室開發方法的法規來適用這類的法規,所謂的體外診斷器材(IVD, In Vitro diagnostic devices)和實驗室開發方法(Lab developed test)是有一些不太一樣的地方,實驗室開發方法通常是那些進步快速的領域之檢驗項目,因為變化太快速了,要是走IVD法規路線,可能核准完市場已經變化且技術同時已經落後,這部分在次世代定序領域尤為常見。

前陣子其實有蠻多風波的特管辦法(特定醫療技術檢查檢驗醫療儀器實行或使用管理辦法),就是把台灣實驗室開發方法納入管理,其中很多關於資格和操作細節都引發各方人馬的意見。回到臨床次世代定序,因其複雜的特性使其很難以IVD的模式通過,直到如今,美國市場上只有四個腫瘤為主的NGS檢驗項目:
- Oncomine Dx Target Test: 2018/10/17
- 包含46個腫瘤相關基因檢測
- MSK-Impact: 2017/11/15
- 包含468個腫瘤相關基因檢測
- FoundationOne CDx: 2017/11/3
- 包含324個腫瘤相關基因檢測
- Guardant360 CDx: 2020/08/07
- 第一個液態活檢針對55個腫瘤基因
體外診斷器材的查驗,其中牽涉到從技術研發、臨床前試驗、第I、II、III期臨床試驗、上市前審查到衛福部查驗登記以及上市後檢測,相對在實驗室自行發展檢測方法(LDT)中,通常是以實驗室的品質系統來取代臨床前試驗、第I、II、III期之臨床試驗,所以相對來說,彈性較大,但是實驗室本身的管控就變得非常重要。
目前在臨床實驗室認證台灣非常重視的CAP(College of Amierical Pathologist)的規範,其近期也有一系列文章和網站資源在提供次世代定序的實驗室規範,蠻值得一看的。
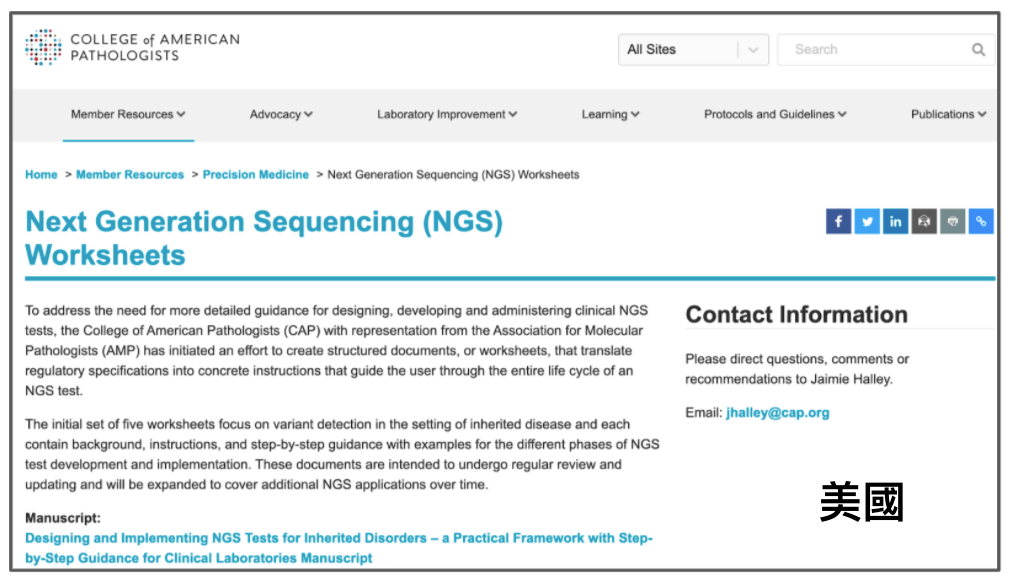
其中主要是分別在Test Content Design, Test Optimization, Test Validation, Quality Management, Bioinformatics and IT這幾個方向有建立一個方便實驗室追尋的worksheet。

在這邊我們同時來比較一下台灣這兩年衛福部提供的指引來探討一下異同和改變,也剛好是前陣子在科內給的演講。

簡報的左邊是引用來自xkcd的卡通圖,談的是當代操作系統如Linux(當今所有雲端系統基本上都基於linux核心,再往上疊加),完整的計算機操作系統,奠基在非常多開發者之程序中,有的程序寫得很好,有的則是很脆弱,假如沒有對整體有個巨觀,很可能出錯了都無法抓蟲,這讓我聯想到次世代定序作為檢驗服務給我的感覺(也是聽完郭靜穎老師和蘇剛毅老師在臨床次世代定序中關於確效認證等等的主題後的想法),雖然目前臨床次世代服務中,所牽涉到的許多細節其實已經靠廠商簡化許多,比如機台自動化、檢體前處理優化到分析套裝軟體,但依舊是複雜模塊的堆疊,如同這左邊這張圖。
台灣這幾年政府也希望能回應醫療人員對於次世代導入臨床之需求,所以分別在2020年和今年年初都有分別針對遺傳類和腫瘤類的基因檢測給予相關的指引,這邊往下比較台灣指引在20200811年公告之精準醫療分子檢測實驗室檢測技術指引-是世代定序應用於遺傳類疾病檢測及20210311公告之精準醫療分子檢測實驗室檢測技術指引-是世代定序應用於腫瘤檢測(草案)。
的確在業界常聽到大家抱怨台灣法規在次世代定序臨床易用這塊很緩慢,也導致普遍醫學中心外並沒有多少人應用次世代定序來幫忙民眾,相對於中國來說,其在2018年開始就有第一份的临床基因检测报告规范与基因检测行业共识探讨來,不過樂觀一點,台灣這一兩年陸陸續續也把遺傳類和腫瘤類指引提出,所以這邊就這個指引和美國CAP的內容來相對應一下,也可以看到今年3月的指引比去年的遺傳類檢測的看法更加純熟。


比較兩個台灣的指引,可以看出今年3月提出的版本,對於生物資訊流程的部分有更多著墨,尤其是在基因資料庫使用與管理建議和檢測報告格式,這部分仔細檢討其實也是當前台灣臨床實驗室比較弱的部分,整體上兩個指引對照CAP下都有所謂的適用範圍、檢測設計考量、檢體類別、影響檢測因素及檢測個步驟,另外,在腫瘤這份也有多著墨一下變異偵測的部分。

就如同這次課程介紹的,次世代定序的應用非常廣,所以兩份指引都有開宗明義定義說適用的範圍,在不同情況下就會有不同的狀況,所以兩者面向的一個是偏產科和兒科以及成人癌症部分的次世代定序檢測。

往下去看兩份指引在步驟建議上,腫瘤檢測的部分可看到觀念比較全面,也加入分析後的品控想法,也有去討論基因庫定序之複雜度及潛在風險評估的部分。

在檢體處理部分,其實跟CAP的工作單中描述的大概相同,但台灣這邊的品質範圍拉的比較大一點,規範細節相似。

檢體在建庫部分也希望臨床檢測的SOP中,也要記錄清楚建庫所相關的細節像是製備方法(擴增法或是捕捉法)、建庫流程中針對核酸片段放大之效能及允收標準,也提到品管物質,這部分的確是目前臨床實驗室比較難符合的地方,同時也會增加品控的成本。

最後在生物資訊分析流程中也希望能把整個流程中所處理的流程、數據分析方法及變異點偵測邏輯都要詳述,且所使用的軟體和資料庫版本都有有所談論,這幾天在多數臨床實驗室沒有配置生資人員情況下,大都是依賴廠商,廠商有時候也是代理國外軟體,所以造成細節部分都沒有人搞得清楚。

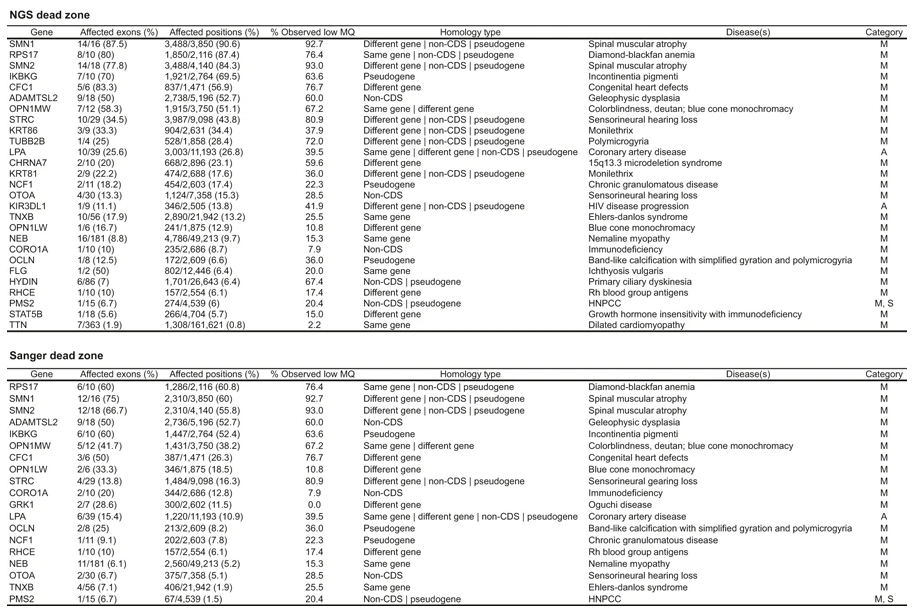
進入到檢測品質這邊,跳過檢體和DNA品質這塊,內容跟前述的部分雷同,這裡也有提到要詳述定序覆蓋率,不是只描述平均覆蓋率而已,還要去描述最低和最高的部分,以及目標區域的定序深度(目前臨床使用都還是Panel為主,如同陳沛隆醫師所講),另外,同行和異形合子的基因頻率也要去描述其篩選條件。

取得定序機台的read後,必須要把相關評估read品質和篩選之流程都要記錄清楚,且前一步驟中是否有去掉序列也要描述,最後其比例和重複定序片段的數量和百分比都要紀錄(在此堂課郭老師給予的測試數據中,也會發現其重複定序的片段比率偏高,也是要去理解背後的因素,是否有非預期的原因所造成)

同時整個流程中的定位也要去看其相關指標的細節,不過這算是所謂的post alignment quality control的部分,要去看reads拼貼的狀況,畢竟最後變異的分析都是基於這些read alignment後的結果)。

最後變異偵測的參數則是要依據臨床場景有不太一樣的想法,在做遺傳類變異和腫瘤類體細胞變異偵測上就是不太一樣,其中變異等級之指標、整體變異的指標、變異的變異頻率都是重要需要紀錄的資訊。

變異註解的部分也是一個大功夫,郭老師也花費一整堂的部分來描述這流程中要考慮到的細節和需要調用的資料庫,中間所使用的資料庫種類版本,以及流程都是需要系統性標準化的,另外,判讀後的結果也要好好記錄下來。

最後檢測報告該如何撰寫要記錄什麼,在精準醫療分子檢測實驗室檢測技術指引中也有大概的介紹,大體是該包含什麼資訊:位點、臨床註解、檢測方法、相關限制。

在腫瘤檢測的指引中,開宗明義希望報告儘可能剪短、實際,這中間就給予個別實驗室一定的空間來決定,比較重要的部分則是所謂的陰性結果(Pertinent negative)的呈現,相對來說,就是臨床上喜歡的“rule-out”思考邏輯。
整體來說,次世代基因定序涉到多個流程,每個流程之間都要有相對應之品質管控,從檢體前處理、建庫、定序初始資料、生物資訊流程和報告,如此複雜的過程其實“相當富有挑戰”,但也是吸引人投入的地方吧!也期待越來越多人投入這個領域,能讓這樣技術解放更多關於人們的生理資訊,一方面讓臨床更能給予民眾幫助!