這篇接續精準醫學邀請演講:臨床定序技術與分子診斷-由短到長、一代到三代 (ㄧ)的內容。

上一部分大概分享到了病生理學的部分,從單位點的變化,要開始往下去分享一些人類基因序列的模式。
短串聯重複(Short Tandem Repeats)和微衛星(Microsatellites)
短串聯重複(Short Tandem Repeats,簡稱STRs)和微衛星(Microsatellites)這兩個在形容基因組結構的名詞,其實本質上是一樣的,但在體細胞談論遺傳時,都是以短串聯重複STRs為主,而在腫瘤領域在探討這類短片段重複的序列特性,則是在。它們都指的是一段由短的核苷酸序列(通常為2-6個核苷酸)構成的重複單元在基因組中連續出現的區域。
STRs的特性其實是其重複單元會造成DNA複製過程中的滑移(slippage)現象。當DNA聚合酶在複製過程中失去與模板DNA的配對時,可能會引起滑移,導致重複單元的增加或減少。這使得STRs在不同個體間具有高度的多態性,為遺傳學和人類遺傳病的研究提供了重要的信息。
由於STRs的多態性,它們在生物學和醫學上具有多種應用。在法醫學中,STRs被廣泛用作DNA指紋圖識別,用於確定遺傳親子關係、個體辨認以及犯罪現場物證分析。在人類遺傳病研究中,部分STRs與特定的遺傳病相關,如亨廷頓舞蹈症和類固醇21-羥酸缺乏症等。
然而,STRs在基因組中的功能仍然不完全清楚,如同前面一篇文章有分享到其實到2022年才有第一個完整的人類基因參考序列,那當然這類複雜基因序列在族群中的特性才開始能慢慢去理解。有研究表明,某些STRs位於基因的調控區域,可能影響基因表達,而其他STRs位於蛋白質編碼區域,可能影響蛋白質結構和功能。隨著對基因組結構和功能的進一步研究,STRs在遺傳學和基因組學領域的作用和重要性可能會得到更多闡釋。
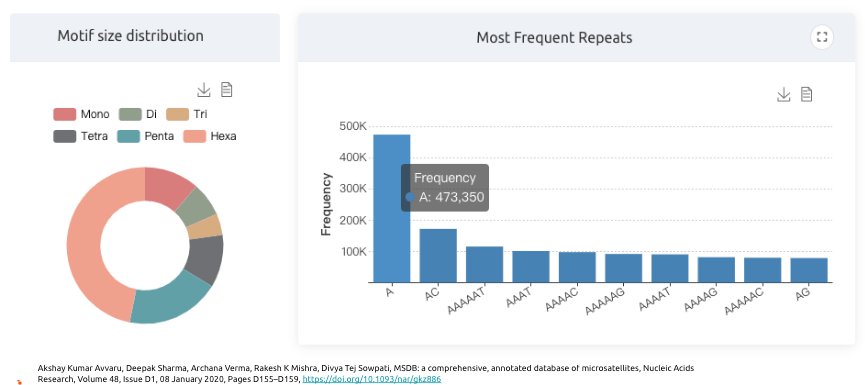
從這邊文獻中,可以看到一些有趣的數據,比如STR目前定義是1-6bps的重複片段,而從這邊可以看出來,主要以6bp的重複片段所佔的比如最高,其中又以A是最常見的序列。
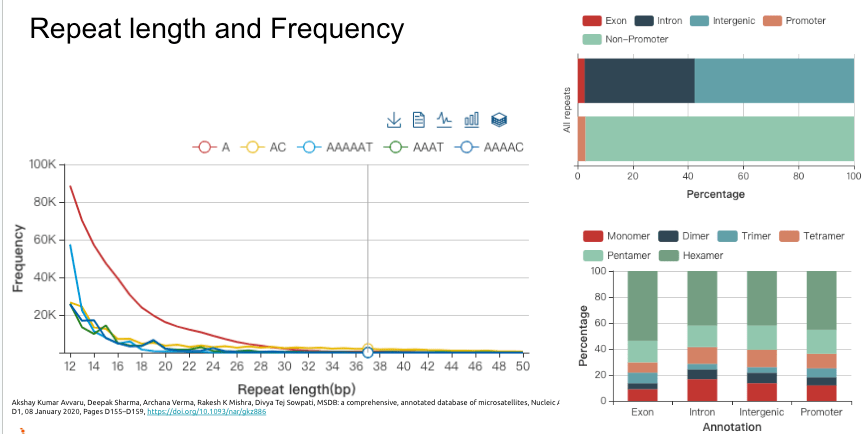
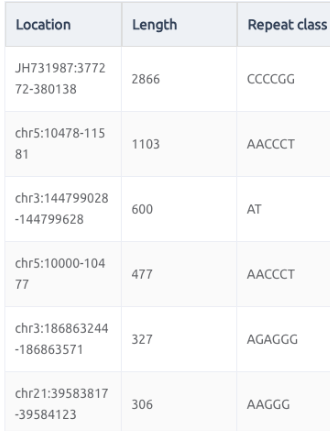
從上面的統計圖表,可以知道最常見的重複數量大概是在30次以下,位在外顯子上的比例是最小的,最多的就是在飛機因區域,再來則是基因內的內顯子區域。不過有重複序列可以到2866次,這個重複數量是很難想像的。

而這樣的重複片段在不同基因結構的區域,其實也有不少相關疾病被知道,比如上面圖片來自Hannan, A. J. (2018). Tandem repeats mediating genetic plasticity in health and disease. Nature Reviews Genetics, 19(5), 286-298.文章,便可以看到下面相關的疾病:
短串聯重複(Short Tandem Repeats,STRs)在基因組中具有高度變異性,與某些遺傳性疾病密切相關。以下是幾個與STRs相關的遺傳疾病範例:
- 亨廷頓舞蹈症(Huntington’s disease):這是一種神經退行性疾病,由於CAG三核苷酸重複序列在HTT基因中過多重複,導致胺基酸葡萄糖胺在亨廷頓蛋白中過多積累,進而引起神經細胞功能障礙和死亡。
- 類固醇21-羥酸缺乏症(Congenital Adrenal Hyperplasia):這是一種常染色體隱性遺傳病,與CYP21A2基因的STRs變異相關。當重複序列中的CYP21A2基因發生變異時,可能導致腎上腺皮質激素合成受阻。
- 慢性肌無力(Myotonic Dystrophy):這是一種肌肉失去力量和無法放鬆的遺傳性疾病。在第一型慢性肌無力(DM1)中,DMPK基因內的CTG三核苷酸重複過多,導致異常的mRNA積累,從而影響肌肉細胞功能。在第二型慢性肌無力(DM2)中,則是由於ZNF9基因內的CCTG四核苷酸重複過多。
- 弗里德勒依託病(Friedreich’s Ataxia):這是一種神經退行性疾病,與FXN基因中的GAA三核苷酸重複過多相關。這種重複可能導致鐵依賴型抗氧化酶(frataxin)的缺乏,進一步引起神經細胞和心臟細胞的損傷。
- 極端X綜合症(Fragile X Syndrome):這是一種智力障礙和行為異常的遺傳性疾病,與FMR1基因中CGG三核苷酸重
- 脊髓小腦萎縮症(Spinocerebellar Ataxias,SCAs):這是一組神經退行性疾病,影響脊髓和小腦。多種SCAs與STRs變異有關,例如SCA1(CAG重複於ATXN1基因)、SCA2(CAG重複於ATXN2基因)、SCA3(CAG重複於ATXN3基因)等。
- 肺泡蛋白病(Pulmonary Alveolar Proteinosis,PAP):此疾病與CSF2RA基因中的STRs變異相關。該變異導致肺泡巨噬細胞功能異常,進而引起肺泡內蛋白質過度積累。
- 黑尿症(Alkaptonuria):這是一種代謝性疾病,與HGD基因中的STRs變異相關。這種變異導致同尿苯酸氧化酶(homogentisate 1,2-dioxygenase)的功能缺陷,使尿中的同尿苯酸無法正常代謝,進而導致黑尿和其他相關症狀。
- 骨化性纖維組織病(Fibrodysplasia Ossificans Progressiva,FOP):這是一種極為罕見的遺傳性疾病,與ACVR1基因中的STRs變異相關。該變異導致骨骼肌纖維被骨組織所取代,造成肌肉僵硬和關節活動受限。
- 肺動脈高壓(Pulmonary Arterial Hypertension,PAH):這是一種影響肺血管的疾病,與BMPR2基因中的STRs變異相關。該變異導致骨形成蛋白受體2(bone morphogenetic protein receptor type 2)的功能缺陷,使肺動脈壓力上升,進而引起心臟負擔加重。
拷貝數變異(Copy Number Variants):在長一點的重複序列
上面的短串聯重複片段(STRs),既然叫做“短”,那麼就有在長一點點的重複序列特徵,那麼就會被歸類在所謂的拷貝數變異,歸類在更大範圍的結構變化(Structual Variation),換句話說,拷貝數變異(Copy Number Variants)涵蓋了一個相對廣泛的基因組片段大小範,從數百個核苷酸(bp)到數百萬個核苷酸(bp)不等。這些變異可以是重複(增加拷貝數)或缺失(減少拷貝數)。然而,CNVs的精確大小範圍會根據定義和檢測方法而有所不同。一些研究將CNVs定義為影響至少1,000個核苷酸(1 kb)的變異,而其他研究則將閾值設置為50,000個核苷酸(50 kb)或更大。隨著檢測技術的不斷進步,研究人員現在能夠在更細的尺度上檢測到更小的CNVs,進一步擴大了我們對這些變異的認識和研究範疇。
P.S: 這也是為什麼在理解基因體學的時候,一定要搭配對於技術的理解,很多時候,如同以管窺天,我們就會以為天就是圓的。

圖片來自於Phenotypic impact of genomic structural variation: insights from and for human disease, Nature Genetics Review, 2013
臨床上,用來做這類大片段變異的工具,最常見的就是產前檢查的項目,如基因晶片ArrayCGH等,偏向在新生兒遺傳異常的偵測,但成人上相對就還沒有臨床應用,與CNVs相關的疾病涵蓋了許多不同的領域,包括神經發育障礙、精神疾病、自閉症和其他遺傳病。以下是一些與CNVs相關的疾病範例:
- 神經發育障礙:部分學習障礙和智力障礙與CNVs有關。例如,部分Williams症候群(一種罕見的神經發育病)患者的基因組中,某個區域的拷貝數減少。
- 精神疾病:與精神分裂症相關的多個CNVs已被發現,包括1q21.1、15q11.2、15q13.3和22q11.21等區域的拷貝數變異。
- 自閉症譜系障礙:已經確定了與自閉症相關的多個CNVs,如16p11.2、22q13.3和15q11-13等區域的拷貝數變異。
- 遺傳病:某些遺傳病也與CNVs有關,例如Charcot-Marie-Tooth病(一種遺傳性神經病)中,17p12區域的拷貝數變異與疾病的發生有關。
- 免疫缺陷病:部分免疫缺陷病也與CNVs相關,如DiGeorge症候群(一種罕見的免疫缺陷病),在22q11.2區域的拷貝數減少與疾病的發生有關。
- 癌症:在某些癌症患者中,已經觀察到了特定基因的拷貝數變異。例如,乳腺癌中的ERBB2(又稱HER2)基因放大與腫瘤的惡性程度有關。
這邊其實也會觀察到有些複雜的疾病表型,如小胖威力Willian syndrome,就有多種可能會造成,這也代表者疾病定義在未來可能會需要調整和精進。

上面這張圖,則是展現了已知CNV在人類基因組上的分佈情況,另一方面,也暗示了不同人類染色體其上基因序列複雜度也是不太一樣,性染色體、染色體22號、染色體16號、染色體15號、染色體9號等等都是相對來說發現比較多拷貝數變化的染色體。

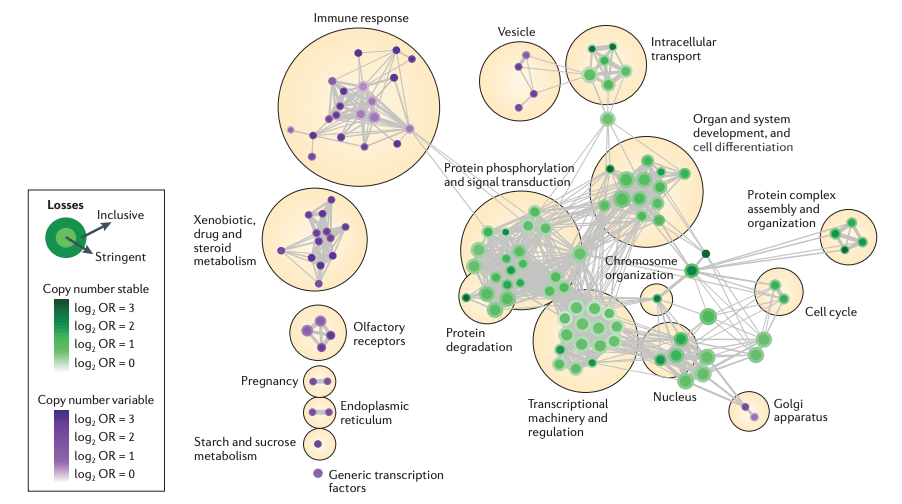
上面的圖片則是進一步介紹不同CNVs在特定功能區域的分佈,很明顯可以看到在non-coding基因區域、CpG island、Promoters都是比較多的,暗示者其在參與基因調控上扮演的角色較重。下面的分析則是進一步用CNVs所在區域的已知功能,做關聯性的分析,似乎也可以看到更比較複雜的生物功能有關,如免疫、染色體結構等等。
更複雜的3D 基因體結構
你以為基因體學就停在對於序列的模式和異常嗎?其實人類越探索,就越發現其中的複雜,最近幾年因為基因定序的進步,認為染色體在細胞核中的折疊和排列,本身就會對於基因產生複雜的調控。這領域稱作3D基因體學(3D genomics),或是空間基因體學,是一個新興的研究領域,旨在研究基因組在三維空間中的組織和折疊結構,以及這些結構對基因調控和基因組功能的影響。
3D基因體學的主要概念包括以下幾個方面:
- 染色體領域(Chromosome Territories):在細胞核中,不同的染色體被發現佔據了自己獨特的空間區域,稱為染色體領域。這些領域有助於維持基因組的穩定性和遺傳信息的整合。
- 染色體組織域(Chromatin Domains):染色質被組織成一個分層的結構。在這個結構中,相鄰的基因區域可能因為染色質折疊而相互作用,從而促使協同調控的基因共享調控元件。
- 開放染色質區域(Open Chromatin Regions):開放染色質區域是指在基因組中易於轉錄因子和其他調控因子結合的區域。這些區域通常與基因的啟動子、增強子或其他調控元件相關。
- 染色質互作(Chromatin Interactions):基因組中不同區域之間會發生物理接觸,這些接觸有助於調控基因表達。例如,增強子和啟動子之間的互作可以調控特定基因的表達。
為了研究3D基因體組織,科學家們已經開發了多種實驗方法,如染色體共閘定(Chromosome Conformation Capture,3C)技術及其衍生技術(如4C、5C和Hi-C)。這些方法允許研究人員獲得基因組在三維空間中的組織信息,並揭示染色質互作和基因調控的機制,這些方法目前都是停留在研究技術,當然也代表者離臨床距離較遠,但這代表我們必須理解從基因到表型的距離,還牽涉到這麼多東西。
隨著3D基因體學研究的深入,有很多這領域的發現和提出的概念,當然,要知道的就是這些概念都是伴隨者檢驗技術所生的,必定有其侷限之處:
- TADs(Topologically Associating Domains):基因組中的特定區域在三維空間中被發現存在高度自我聯結的結構,稱為TADs。TADs的結構與基因調控密切相關,因為它們使得調控元件(如增強子)能夠更容易地與目標基因接觸。
- 染色質環(Chromatin Loops):在染色質折疊的過程中,某些區域(如增強子和啟動子)被拉近並形成物理上的接觸,形成染色質環。這種結構有助於協調基因表達和精確地調控基因活性。
- 細胞類型特異性的3D組織:不同細胞類型之間的基因組組織可能存在差異。這些差異可能影響基因表達的調控,並有助於維持細胞特性和功能。
- 3D基因體學與疾病:基因組組織的改變可能導致疾病。例如,某些癌症中的染色質重排可能導致調控元件錯誤地與目標基因互作,從而導致基因的過度表達或失活。
總之,3D基因體學是一個新興且快速發展的研究領域,它對我們理解基因調控和基因組功能有著重要的意義。雖然目前還在探索跟疾病的關聯,但這些是知道跟基因組立體結構相關的疾病:
- 癌症:某些癌症中的染色質重排可能導致調控元件(如增強子)與錯誤的目標基因互作,從而導致基因的過度表達或失活。例如,某些白血病(如急性淋巴細胞性白血病)與基因重排導致的染色質環異常有關。
- 神經發育障礙:染色質環和TADs在神經發育過程中的基因調控中也起著關鍵作用。某些神經發育障礙(如自閉症、智力障礙和精神分裂症)與這些結構的異常有關。
- 先天畸形:某些先天性疾病,如林茨(Limb-Body Wall Complex,LBWC)綜合徵和菲林-麥克德蒙德(Feingold-McKusick)綜合徵,與基因調控元件(如增強子)在染色質環或TADs中的異常定位有關。
- 罕見遺傳病:某些罕見遺傳性疾病,如Facioscapulohumeral Muscular Dystrophy(FSHD),也與染色質環或TADs的異常有關。FSHD與染色質結構中D4Z4重複序列的減少有關,這可能導致調控元件與錯誤的基因互作,進而引起疾病。
閱讀參考
跟拷貝數變異相關的文獻
- Redon, R., Ishikawa, S., Fitch, K.R., Feuk, L., Perry, G.H., Andrews, T.D., Fiegler, H., Shapero, M.H., Carson, A.R., Chen, W., Cho, E.K., Dallaire, S., Freeman, J.L., Gonzalez, J.R., Gratacos, M., Huang, J., Kalaitzopoulos, D., Komura, D., MacDonald, J.R., Marshall, C.R., Mei, R., Montgomery, L., Nishimura, K., Okamura, K., Shen, F., Somerville, M.J., Tchinda, J., Valsesia, A., Woodwark, C., Yang, F., Zhang, J., Zerjal, T., Zhang, J., Armengol, L., Conrad, D.F., Estivill, X., Tyler-Smith, C., Carter, N.P., Aburatani, H., Lee, C., Jones, K.W., Scherer, S.W., & Hurles, M.E. (2006). “Global variation in copy number in the human genome." Nature, 444(7118), 444-454.
- Stankiewicz, P., & Lupski, J.R. (2010). “Structural Variation in the Human Genome and its Role in Disease." Annual Review of Medicine, 61, 437-455.
- Weischenfeldt, J., Symmons, O., Spitz, F., & Korbel, J.O. (2013). “Phenotypic Impact of Genomic Structural Variation: Insights from and for Human Disease." Nature Reviews Genetics, 14(2), 125-138.
- Zarrei, M., MacDonald, J.R., Merico, D., & Scherer, S.W. (2015). “A Copy Number Variation Map of the Human Genome." Nature Reviews Genetics, 16(3), 172-183.
跟3D基因體學相關的文獻
- Dekker, J., Marti-Renom, M. A., & Mirny, L. A. (2013). Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nature Reviews Genetics, 14(6), 390-403.
- Lieberman-Aiden, E., van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., … & Dekker, J. (2009). Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science, 326(5950), 289-293.
- Dixon, J. R., Selvaraj, S., Yue, F., Kim, A., Li, Y., Shen, Y., … & Ren, B. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature, 485(7398), 376-380.
- Rao, S. S., Huntley, M. H., Durand, N. C., Stamenova, E. K., Bochkov, I. D., Robinson, J. T., … & Aiden, E. L. (2014). A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell, 159(7), 1665-1680.
- Bonev, B., & Cavalli, G. (2016). Organization and function of the 3D genome. Nature Reviews Genetics, 17(11), 661-678.