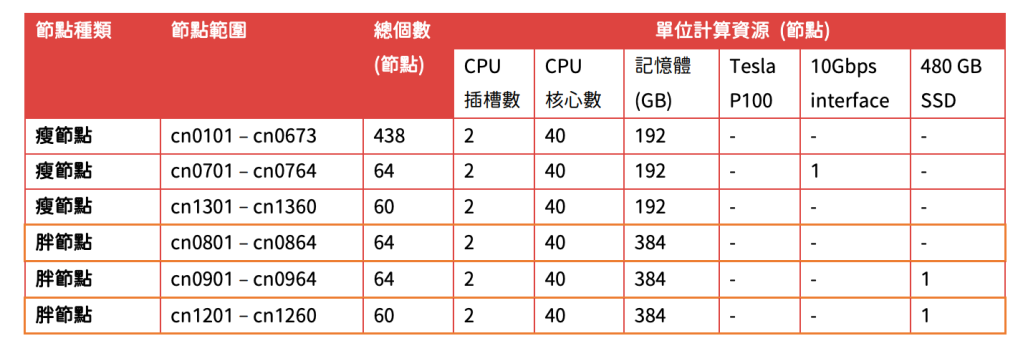
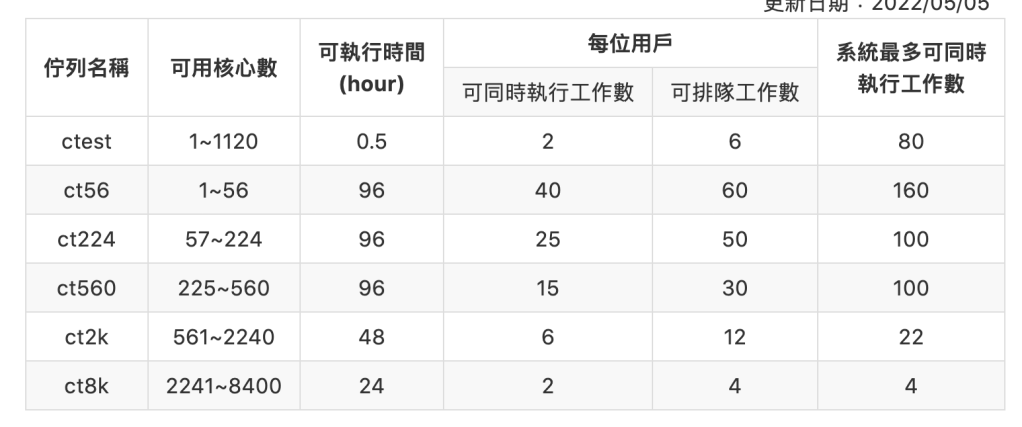
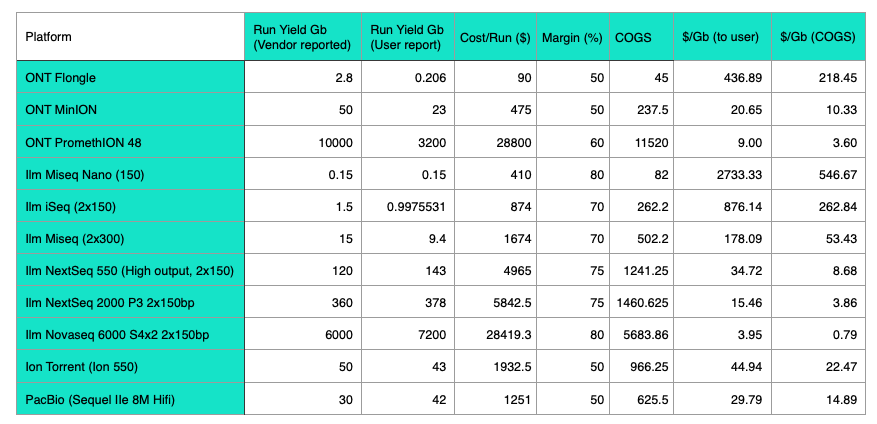
最近在Twitter上看到Ming Tommy Tang的分享關於生物資訊學習資源,想說就稍微紀錄一下,這邊也順便把之前寫過的一些關於資源類的文章匯集一起。
次世代定序資料分析學習資源(NGS analysis learning material)
這邊是原始的Twitter連結,我們把內容轉化為中文資訊,方便有興趣的人可以搜索得到。
由哈佛大學生物統計教授Rafael Irizarry所建立的HarvardX Biomedical Data Science Open Online Training,內容涵蓋由淺入手,由理論到實務,由桌面端到雲端,主要是以R和Python為主。
- Data Analysis for the Life Sciences Series
- Statistics and R
- Introduction to Linear Models and Matrix Algebra
- Statistical Interference and Modeling for High-throughput Experiments
- High-Dimensional Data Analysis
- Genomics Data Analysis Series
- Introduction to Bioconductor: Annotation and Analysis of Genomes and Genomic assays
- High-performance computing for reproducible genomics with Bioconductor
- Case Studies in functional Genomics
- Using Python for Research
猶他大學的人類基因體學教授Aaron Quinlan,本身是bedtools作者,他的課程Applied Computational Genomics,有教學影片和相關簡報。這邊可以稍微看一下他的上課大綱:
- Course Overview and Intro to Unix
- Pattern searching in the human genome
- Data frames and Importing Data
- Intro to the tidyverse
- DNA sequencing technologies
- FastQ format and tools
- Sequence mapping and alignment
- Samtools and IGV
- Poisson Processes in Biology
- An introduction to awk and bioawk
- Genetic Variation
- SNP and INDEL discovery
- Rates and patterns of human germline variation
- VCF format, Hardy Weinberg Equilibrium, VCF toolkits
- VCF annotation and interpretation
- Genome Annotation and Resources
- Genome Annotation Formats
- Genome arithmetic with bedtools
- Monte Carlo simulations and more on UNIX
- Descriptive plots. The Central Limit Theorem
猴子演算法課程Bioinformatic Algorithms,是由Carnegie Mellon University教授Phillip Compeau(本身也創辦Rosalind,有點像是生物資訊領域的leetcode)和UCSD教授Pavel Pevzner所一起規劃的,其內容有影片、Coursera課程、電子書、互動式網頁等等不同的媒介來學習,主要針對生物問題展開,並且討論背後所衍生的演算法,算是很棒的學習素材,另一方面,也能理解生物資訊的重點不是只有程式,而是如何問問題和做假設:
- Origin of replication
- DNA patterns related to Molecular Clocks
- Assemble Genomes
- Sequence Antibiotics
- Compare Biological Sequences
- Fragile Regions in the Human Genome
- Which Animal Gave Us SARS
- How Did Yeast Become a Wine Maker
- How Do we Locate Disease-Causing Mutation
- Why Have Biologists still not developed an HIV Vaccine
- Was T. rex Just a Big Chicken
Biostar創辦人Istvan Albert教授其所規劃的一系列電子書和課程,相對上面的資源來說,算是比較輕量等級的素材,內容定位在淺顯實用。
- The Biostar Handbook
- The Art of Bioinformatics Scripting
- RNA-Seq by Example
- Corona Virus Genome Analysis
- Biostar Workflows
約翰霍普金斯的教授Michael Schatz 其所開設的Computational Genomics : Applied Compaative Genomics,在2018年的版本有提供簡報,最近兩年的則是有提供課程大綱和推薦閱讀。
- Genomic Technologies
- Whole Genome Assembly
- Whole Genome Assembly and Alignment
- The human genome and intro to long reads
- Genomics in the Cloud
- Read mapping
- Variant Analysis
- Structural Variant Analysis and Pangenomics
- Genome Arithmetic and Plane Sweep
- Machine Learning Primer
- Functional Analysis
- Human Evolution
- Huan Genetic Disease
- Cancer Genomics
- Microbiome and Metagenomics
- Genomic Futures
麻省理工學院計算生物學領域的教授Manolis Kellis則是開始蠻多機器學習/深度學習在生物醫學應用的主題,內容以啟發為主,細節則是依賴課後的閱讀,其涵蓋範圍相當廣泛。Github連結