一年一度的Oxford nanopore大會又來了,假如時間不夠,可以直接聽牛津孔洞的CEO James Clarke的演講Update from Oxford Nanopore Technologies,基本上就會是今年他們火力集中的部分,大部分會議中的演講,會慢慢在他們官方的youtube頻道釋出。
簡單來說,奈米孔洞定序是一種無標記、單分子高通量的定序技術,雖然目前開始有越來越多以孔洞技術為基礎的定旭公司,但牛津奈米孔洞公司是其中的市場領頭羊,且整體使用者數量是最多的。
這三年ONT的發展方向越來越令人興奮,絕對是一個值得花心思理解的技術線,總結今年最核心的是:
- 專注Duplex定序的改善:
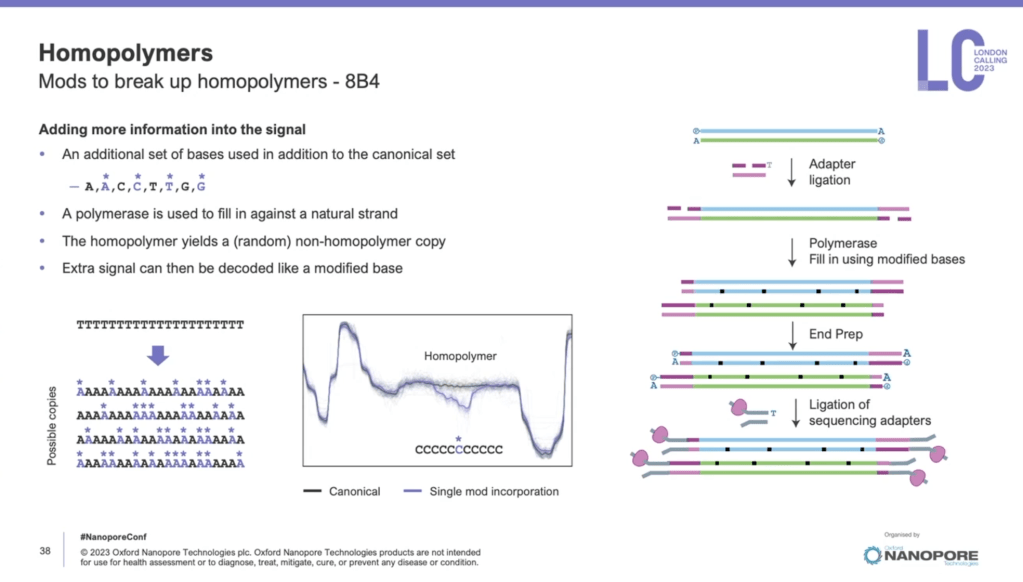
- DNA定序時候的狀況都是雙股的,但之前的定序並沒有強調雙股都會在同一次定序到,而duplex則是同一個雙股分子,兩邊都會被拉進去孔洞,將雙股的資訊互相整合,大大提高定序的正確率,尤其是如多個單序列重複狀況(Homopolymer)。
- 更輕鬆使用的線上分析軟體EPI2ME
- 如何有效分析高通量定序產出的資料一直是個挑戰,因為大部分團隊很難同時擁有生物資訊分析人才,所以ONT一直以來有一個EPI2ME的工具,幫助科學家較輕鬆的擁有一個圖形化介面的分析平台。
- 提高邊緣運算的可近性
- Dorado軟體,相容於Nvidia GPU晶片和Mac M1系列的晶片
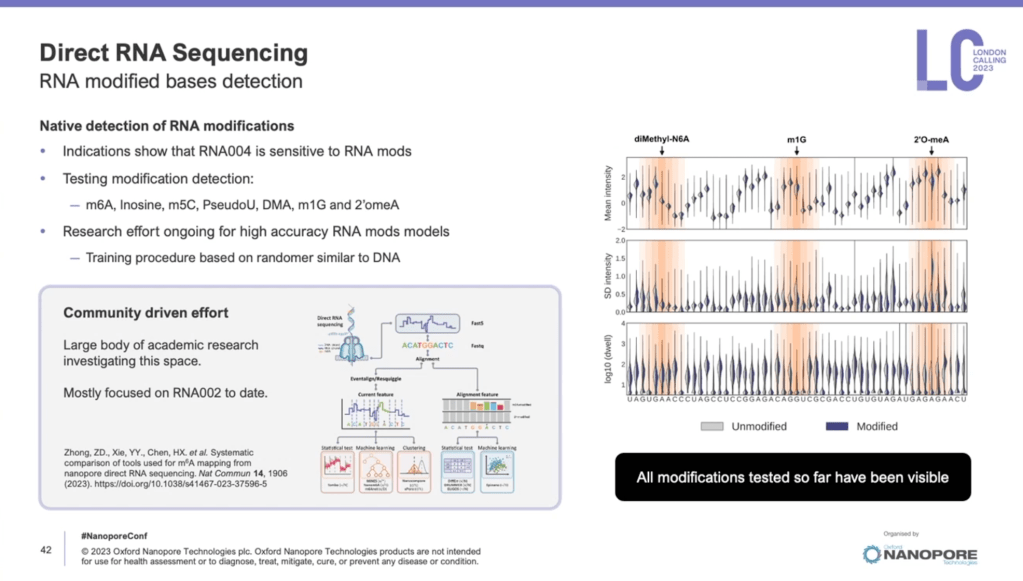
- 直接RNA定序
- 邊緣定序硬體的推出
- 跟iPad整個的定序儀
- MKID的推出(更新原本最入門的定序儀)
- 自製晶片ASIC的優勢
- 透露未來定序設備將更便宜,因ONT是少數核心晶片是自己設計,而非使用現成的模組。
什麼是雙股定序duplex sequencing?
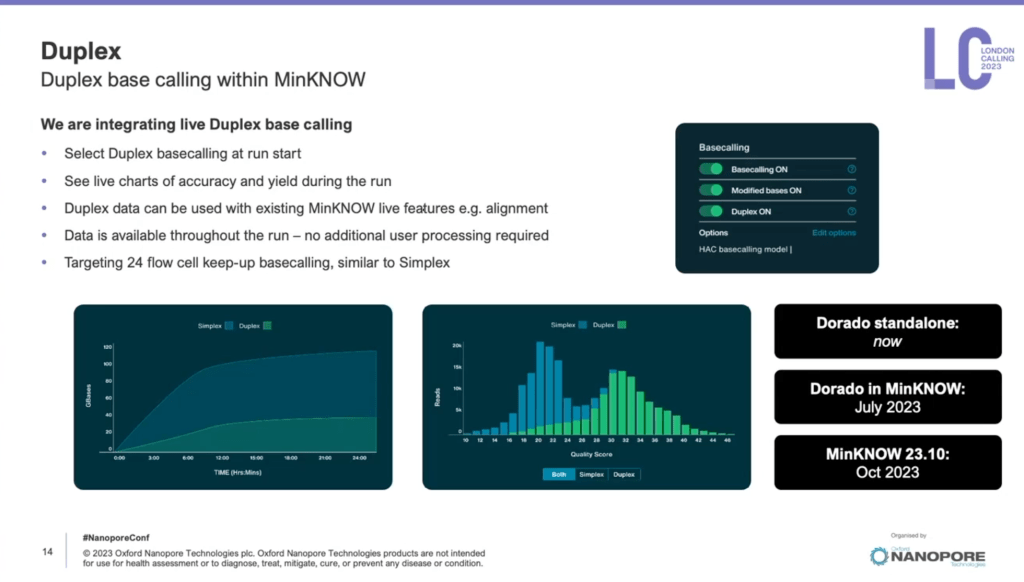
下面的圖示算是一個標準的oxford nanopore定序示意圖,通常我們的定序標的核酸,頭尾會添加上去引子(adaptor),相對於illuminia系統來說,ONT的引子是有蛋白質的,正常情況下,其中一股會被推進去孔洞中,穿過時候產生的電訊號,就會跟其本身的A、T、C、G有關,在使用深度學習的方法轉換成序列。所以在這個狀態下,其實序列是只讀過一次,但後來ONT團隊發現有大約1.5%比例的訊號,有觀察到互補電訊號,應該是發生一股讀完後,另一股也被拉進去,如同下圖的樣子。

這樣讀兩次的現象似乎是一個可以往下發展的模式,用來優化定序的正確率,因為同一個地方被讀了兩次,要是用這兩個互補電訊號來做序列轉換,其正確率應該是可以提高,所以ONT團隊針對相關的細節優化發現可以提高到30%,光針對引子的調整就大幅提高這比例。

這塊的序列轉換就必須使用新的方法學,其實在去年年底有相關分享,不過這個duplex的概念其實是ONT之前1D2的方法學專注的地方,換句話說,他們耕耘過蠻長的時間,只是針對策略再調整。

目前在他們優化的試劑和晶片下,可以得到相當不錯的duplex比例!

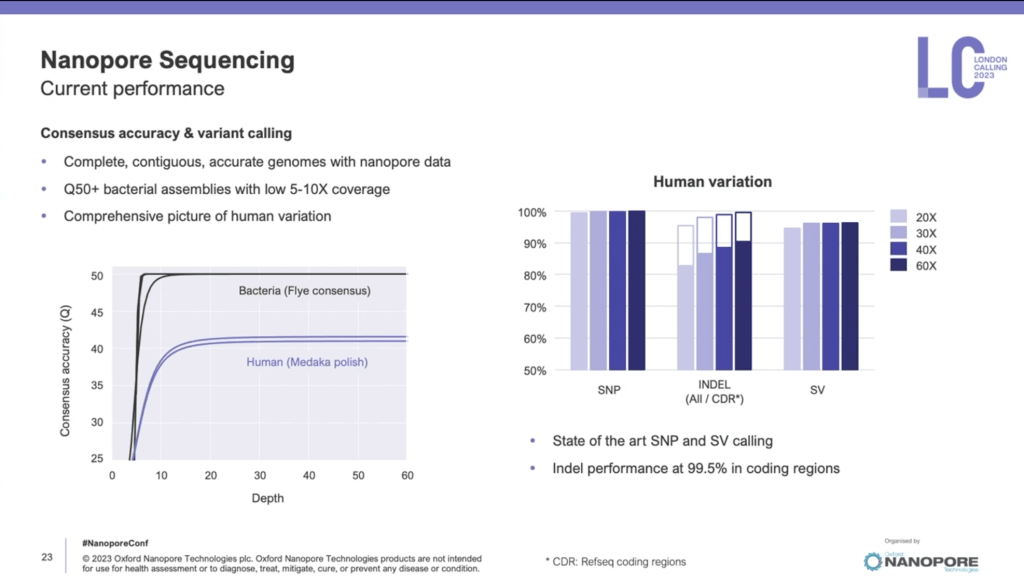
這樣策略下,就能大幅提高定序序列的原始資料正確率,可以看到他整體的品質分數分佈都改變了,就如有常常做就會知道大部分原始資料的高點都會在20左右,所以這個對於品質分佈的提升,其實蠻恐怖的,基本上,可以想成是擁有illumina等級的正確率,而且這個正確率是獨立於序列長度的,不會說你這條讀長越長,其錯誤率越高,這也歸功於ONT並非使用光化學的方法學,會在過程中對於核酸多少有損傷,除非核酸斷裂,不然其對於DNA本身不太會有影響,另外,使用新的模式stereo轉換basecalling,其時間基本上跟之前差不多,不會因此就需要花費更多時間。從下面的圖,也可以看到最新的試劑kit14,其實在單純的單股simplex定序其正確率也有大幅上升,所以這樣的正確率真的很棒。

下面的資料則是在更長的讀長下,其品質分數和準確率間的分佈,在測試中,最長的完美讀長大概可以有40000 bp,而品質達到Q40的讀長則有達到130000bps,整體正確率可以大於99.9%,平均品質分數可以大於Q30。

..
.

.
.
.
.

.

.

..
.

.

..
.
針對一般模式定序在kit14下的表現

.
.
.
.

..

..

.

.
選擇性定序在P2機台

.

.
.

.

.

..

.

.
.

.
.
..

.

.
.
.
.

.

.

.
.

.

.

..
.
.

.

.

.

.
.

.
.

.
.
.
.
