延續上一篇:使用國網中心超級電腦台灣衫建置定序分析流程(一)的內容,往下去記錄到伺服器裡面的一些小細節。目前國網中心裡面的三組超級電腦系統,其實可以發現在規劃上的一些差異性,可以因此用來作為要在哪邊運算的考量點。
思考整體流程的改動
當開始使用高級電腦系統時,其實就會在現有的流程中,多了一段在伺服器的過程,這樣就代表要重新規劃分析的架構和想法,比如哪一個階段上雲,哪一個階段下雲,中間檔案要儲存在哪邊等等的問題。甚至在定序成本降低的時候,可以思考所謂的濕儲存,就是直接把檢體凍起來,有需要再定序。
- 在超級電腦上分析,再將結果轉移到本地桌機進行最後的視覺化
- 先在本地電腦將資料分類或註釋候,上傳到超級電腦中進行接下來的分析
- 直接把整個分析流程架在超級電腦中,在遠端連進去伺服器來看資料
- 將分析結果串接進去一般商用的雲端服務如google cloud、亞馬遜AWS或是微軟的Azure,方便後續的分享和儲存
規劃平行之架構
- 任務平行
- 在整個生物資訊分析流程中,其實是由不同軟體所組成的,每個軟體對於硬體的需求都不同,如何選擇正確的部分來平行運算,可以大大加速流程又節省資源。以分析三代定序的細菌基因組資料來說,就可以簡單分成幾塊,再根據特性來安排運算。
- Basecalling => GPU需求大
- Assembly => CPU需求大
- Annotation => 資料庫整合需求大
- Genotyping => 彈性分析需求大
- Functional comparison => 匯聚分析結果需要
- 在整個生物資訊分析流程中,其實是由不同軟體所組成的,每個軟體對於硬體的需求都不同,如何選擇正確的部分來平行運算,可以大大加速流程又節省資源。以分析三代定序的細菌基因組資料來說,就可以簡單分成幾塊,再根據特性來安排運算。
- 資料平行
- 分析流程中會有需多的中間產物,怎麼讓平行的程式輸入和輸出的資料能平順的接在一起,或是分散式的共用部分註解資料等等
- I/O優化
- 在本地機器上傳檔案,或是超級電腦間資料傳輸,或是超級電腦與雲端服務商如google cloud、AWS或是Azure的串接,這些都會大幅影響整個流程的時間
所需要的相關背景知識
通常阻擋一般人想要嘗試使用這類超級電腦的其實不是經費,反而使用超級電腦的經費需求遠遠小於你所想像的,1000元就可以處理小的細菌組裝,大概100個檢體了,所以覺得比自己花費維護硬體的門檻還低,主要是知識門檻,這邊是相關需要使用超級電腦的背經知識需求:
- 命令行知識
- shellscript: cd、mkdir、ls、cat、grep、awk、sed、ssh、scp、echo
- 叢集電腦指定語法
- PBS
- Slurm
- 基礎編成知識
- python, R
- 版本管理工具
- git, conda, module
- 基本網路協定工具
- putty,
- 基本伺服器環境
- 節點架構
起手式:登入
- 先在windows或是Mac系統中開啟命令行程式
- 輸入以下指令,利用ssh進入節點
下面是成功登入台灣杉三號的畫面,不同超級電腦的登入畫面都不一樣,上面其實都有蠻多重要使用說明,建議都可以記錄或是好好閱讀一下,比如在台灣杉三這邊,可以看到他會把重要指令介紹,比如超級電腦環境中如何去裝載模組,以及提交需要超級電腦運行的代碼,大概只需要知道這些就可以運作了。
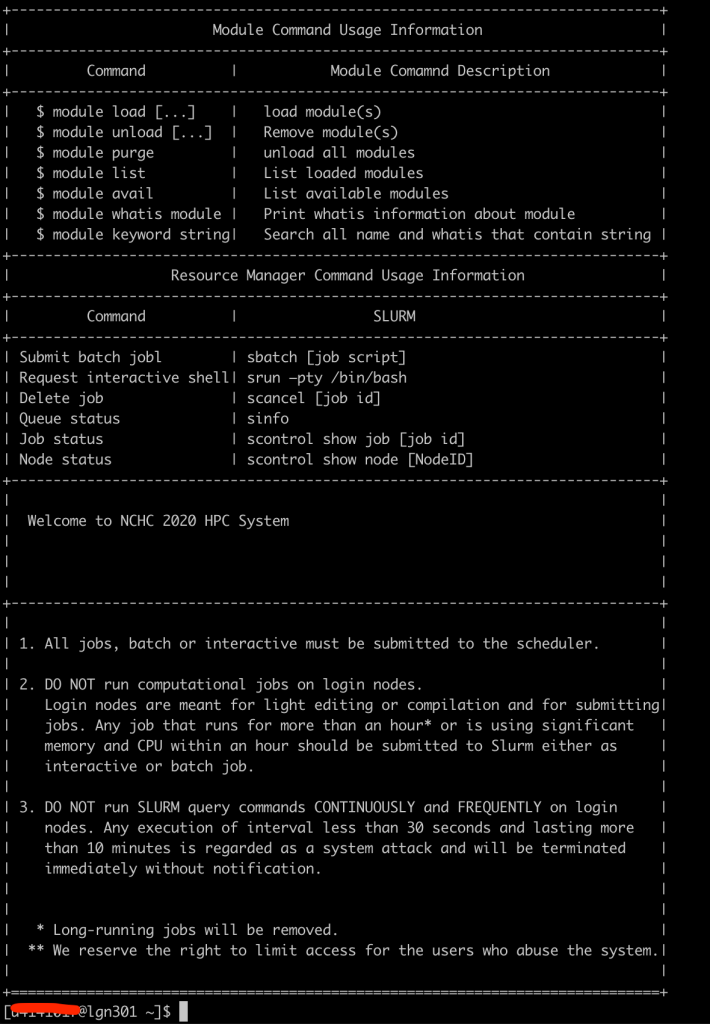
登入後,就可以使用模組系統的指令來看當前環境中有哪些可以使用的軟體,每個叢集電腦去處理隔離環境的方式都不同,這部分可能就是每次使用不同叢集電腦都要特別確認一下。在台灣杉則是使用Environment Modules系統來處理環境變數與全局城程式的管理,Envornment Module算是已有20年歷史在linux系統中幫助管理環境變數的工具。
可以先使用
module available
就會顯示如下的列表,展示當前環境中可以使用的工具,有時候在資料夾結構中有看到自己要用的軟體,在使用module available沒有看到,這時候就需要進一步去聯繫台灣杉這邊的工作人員來幫忙。
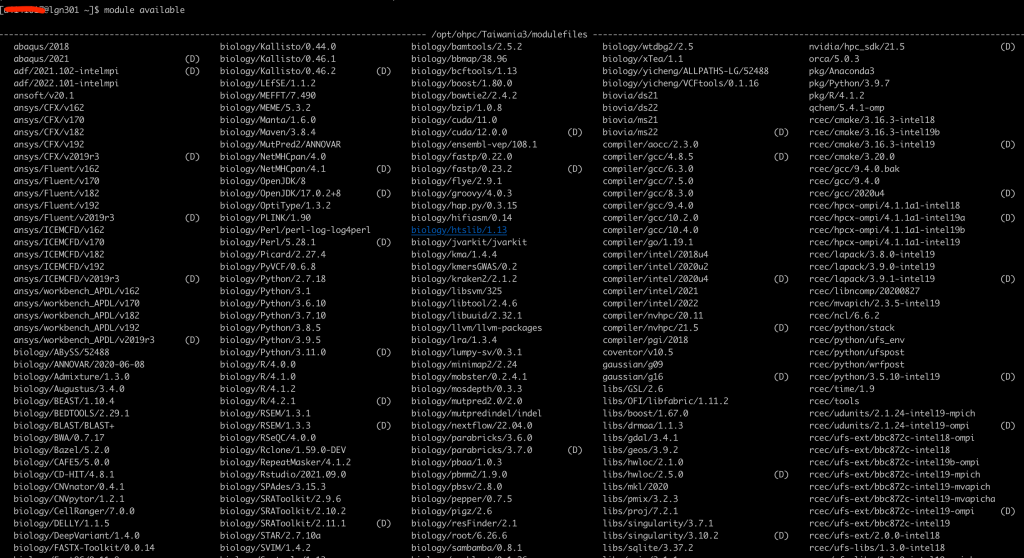
最常見的一個使用場景是使用Anaconda來做軟體版本的管理,這時候就可以先用module load來讀入Anaconda,接者便可以用conda來做環境管理以及安裝軟體,之後將程式丟到運算節點上時候,也可以用這方式來運作。如下的代碼:
# load library with module load
module load pkg/Anaconda3
# able to use conda for environmnenet manage
conda create -n set_upENv
提交代碼至叢集電腦
再來比較讓多數人困惑的是怎麼調用叢集電腦來做運算,其實相當簡單,主要就是提交一份shellscrip代碼,代碼前半部有註明針對叢集電腦運算的需求,比如所用的佇列、使用的代號、需要的記憶體、需要的CPU數量等等,提交後,節點電腦則在區分配相關資源用來執行這個shellscrip代碼。但台灣杉一號和三號的叢集電腦指令不太一樣,台灣杉一號是使用比較傳統的PBS系統,而台灣杉三號則是Slurm系統。
PBS版本
# Shell 說明
#!/bin/bash
# PBS 指令
#PBS -l walltime=00:30:00
#PBS -l select=2:ncpus=16:mpiprocs=16
#PBS -N sample_job
#PBS –q ctest
#PBS –P TRI654321
#PBS –j oe
# 程式與指令
cd $PBS_O_WORKDIRSlurm版本
# Shell 說明
#!/bin/bash
# Slurm 指令
#SBATCH -A ACD110078 # Account name/project number
#SBATCH -J hello_world # Job name
#SBATCH -p test # Partiotion name
#SBATCH -n 24 # Number of MPI tasks (i.e. processes)
#SBATCH -c 1 # Number of cores per MPI task
#SBATCH -N 3 # Maximum number of nodes to be allocated
#SBATCH -o %j.out # Path to the standard output file
#SBATCH -e %j.err # Path to the standard error ouput file
#程式與指令
module load compiler/intel/2020u4 IntelMPI/2020
mpiexec.hydra -bootstrap slurm -n 24 /home/user/bin/intel-hello這邊比較重要的是兩個,一個是PBS -P參數和SBATCH -A參數,這邊就是用來放計畫編號的,會決定所運行的程式費用,扣在哪一個帳號下面,另一個則是佇列參數PBS -q 和SBATCH -p,這邊則是決定所使用的運行環境資源,下面分別有台灣杉一號和三號的佇列類別。
台灣杉一號
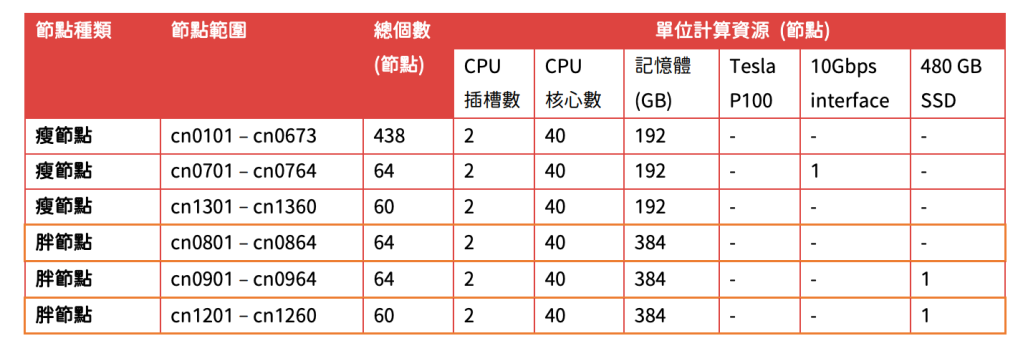
台灣杉三號
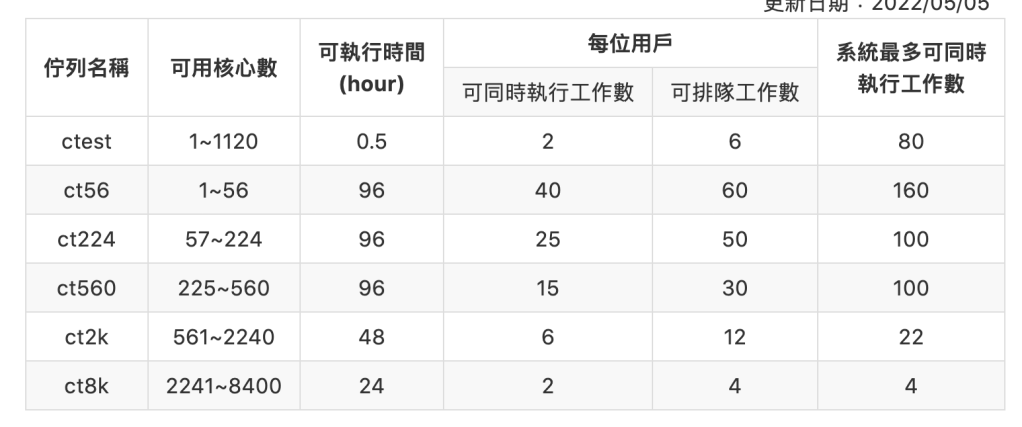
閱讀參考